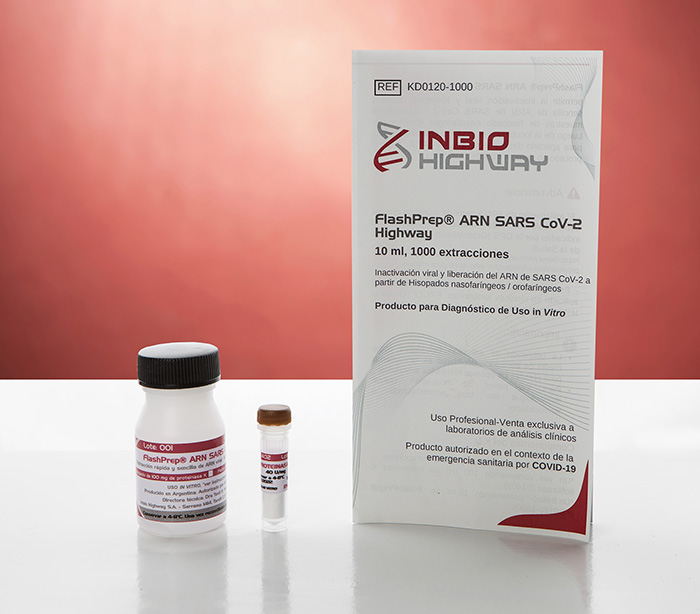
La herramienta para análisis de PCR se llama “FlashPrep® ARN SARS-CoV-2 Highway” y fue aprobada por ANMAT el año pasado. Según describen los investigadores que lo desarrollaron, un operador puede estudiar 94 muestras en 60 minutos en lugar de las 5-6 horas que demandan los métodos comerciales clásicos.
(Agencia CyTA-Leloir)-. Investigadores de la Facultad de Ciencias Exactas y Naturales (FCEN) de la UBA y del CONICET desarrollaron un kit de fácil uso que logra procesar una mayor cantidad de muestras de hisopados para detección del coronavirus SARS-CoV-2 de manera más rápida y económica, según publicaron en la revista con revisión por pares PLOS ONE.
“Nuestra herramienta resultó ser robusta y sensible. Nos alegra saber que nuestra técnica está colaborando con el diagnóstico del virus durante la pandemia”, señaló Valeria Levi, una de las líderes del desarrollo e investigadora del Instituto de Química Biológica (IQUIBICEN) de la FCEN.
La transferencia tecnológica se hizo al ritmo vertiginoso de la urgencia sanitaria. El kit, bautizado FlashPrep® ARN SARS-CoV-2 Highway, lo fabrica y comercializa la empresa argentina Inbio Highway y ya se utiliza en laboratorios de diagnóstico en centros públicos y privados de todo el país.
La nueva tecnología acelera el procesamiento de los hisopados para realizar los estudios de reacción en cadena de la polimerasa en tiempo real, o RT-qPCR: el método de alta especificidad y sensibilidad que amplifica y detecta una región específica del ARN del virus que constituye casi su “huella dactilar”
Según describen los investigadores en el artículo, en lugar de recurrir a una laboriosa extracción previa de la muestra para su análisis, el kit trata el material con una combinación de calor y una proteína (proteasa) de uso habitual en los laboratorios. El procedimiento fue probado de manera exitosa con 106 muestras positivas y otras tantas negativas. Y demostró ser muy rápido: un solo operador puede estudiar 94 muestras en 60 minutos, cuando la misma cantidad requeriría 5 o 6 horas con las técnicas clásicas.
Valeria Genoud, colíder del desarrollo e investigadora del Departamento de Química Biológica de la FCEN de la UBA, destacó la importancia de publicar el desarrollo y las pruebas en una revista tan importante como PLOS ONE, “uno de los mecanismos fundamentales para que el conocimiento generado comience a ser aceptado por la comunidad científica”.
Pero, para los investigadores, también resultó gratificante que su avance no haya terminado solo en un paper. “Fue una gran satisfacción ver cómo nuestro objetivo de aportar una herramienta para colaborar con el control de la pandemia se hizo una realidad”, sostuvieron.
Del proyecto también participaron Martín Stortz, Paula Verneri y Bruno Berardino, del IQUIBICEN, del CONICET y de la UBA; Federico Remes Lenicov y Melina Salvatori, del Instituto de Investigaciones Biomédicas en Retrovirus y SIDA (INBIRS), que depende del CONICET y de la UBA; Ariel Waisman, del FLENI y del CONICET; y Virginia Dansey, de la Unidad de Microanálisis y Métodos Físicos Aplicados a Química Orgánica (UMYMFOR), que depende del CONICET y de la UBA.